上一篇我們的基因體時代-AI, Data和生物資訊 Day03- 基因醫學的數據問題介紹了基因醫學中的數據問題,實際上面對DNA的序列ATCG,我們是在想什麼問題,以及去解析這樣的資料背後所牽涉的複雜架構,我們舉BRCA1這個鼎鼎有名的基因為例,實際去把它的序列部分顯示出來,接者則把目前此領域的專家是如何去處理背後問題,以及基因是由外顯子和內顯子所構成(簡化來說),另外,也分享目前人類兩萬多個基因,其實只有不到十個是人們常探究的,大部分的基因直到目前都還是空白的狀態!
今天我們繼續接者往下深入,為了能把生物問題有效的建構成學習問題,這邊再往下分享一些分子生物學的知識,才會知道我們到底該怎麼利用深度學習來回答這領域的問題。上一篇有分享了一個基因的序列可以分為外顯子(Exon)和內顯子(Intron),本質上這些外顯子是會轉換成蛋白質的序列,一個基因可以經由排列組合不同的外顯子順序和數量,而產生不同的mRNA,進而產生不同的蛋白質,這個現象稱作剪接(splicing),如下圖所示:
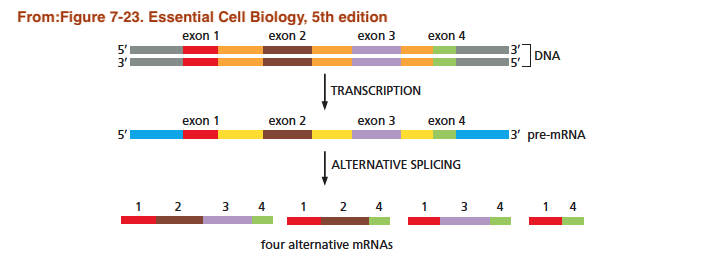
那一個基因中特定的外顯子是否會被剪接呢?就可以把它變成是一個學習問題,而且是一個監督學習(supervised learning)如下: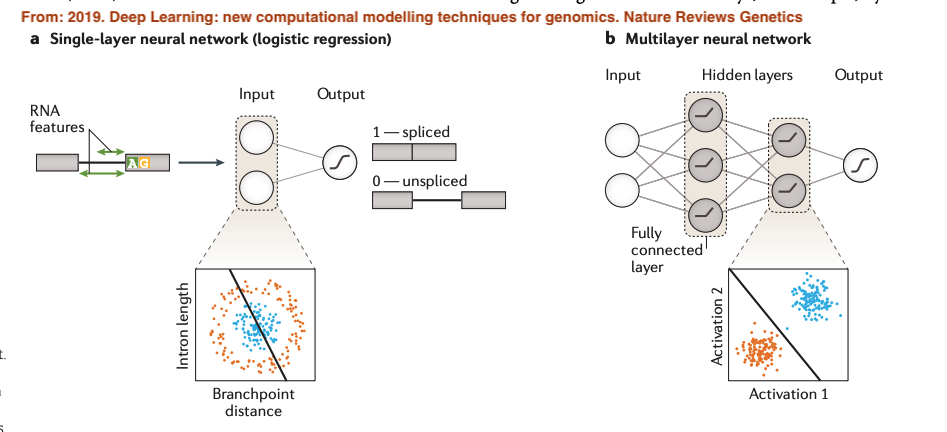
基本上,監督學習的目的是建立一個模型,這個模型有輸入(features),然後會有個輸出(target),在這個問題下,輸入可以是這個外顯子區域的序列,輸出可以是1(剪接)和0(不剪接),而訓練機器模型其實就是在學習他的參數,這過程會最小化所謂的Loss function,這樣才能避免overfitting,也就是能對沒有預測過的資料有較好的結果。
一個基因的外顯子會如何剪接,其實外顯子和內顯子的序列可能會有所影響,如下面這張圖所表示的: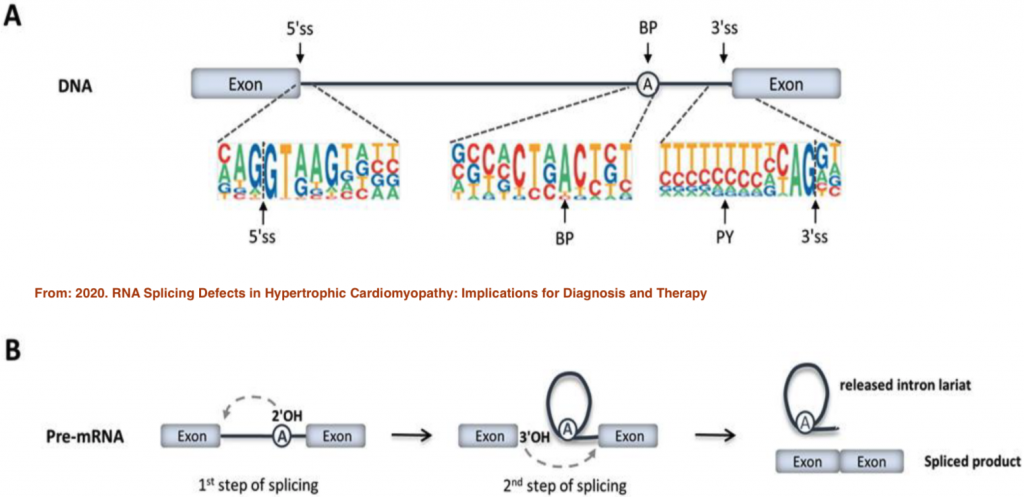
從上圖中,很清楚地展示了外顯子和內顯子交界處會有特徵,另外,兩個外顯子中的內顯子序列,會有個一個區域的序列是會影響剪接的,換句話說,這些都可以轉換成所謂的特徵,來變成輸入的資料。
複雜的資料關聯恰好可以用深度學習的架構來處理,大部分的監督學習的輸入都是表格的資料,也就是一行為一筆資料料,每個欄位是關於這個資料的特徵(feature),當然也有這個資料的標籤,以上面這個預測一個基因上的兩個外顯子會怎麼剪接來說,其區域上的ATCG頻率和內顯子中間區域的ATCG頻率都可以變成輸入表格中的一個欄位,在DNA的序列上,有一個提取特徵的概念叫做k-mer,簡單而言,就是在說一個DNA序列中,在特定長度下去切割這個序列,會產生幾種排列組合,再將這些資訊拿來做進一步的計畫。下面這個就是wiki上面k-mer範例: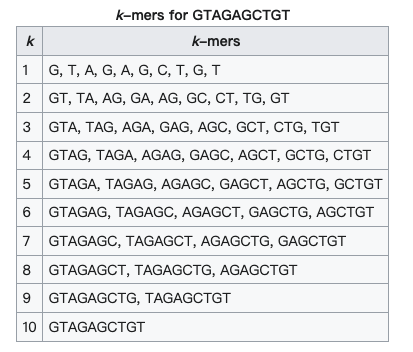
所以簡單來做,我們要將一個分子生物的問題,轉換乘下面這個資料架構,接者就可以來建模: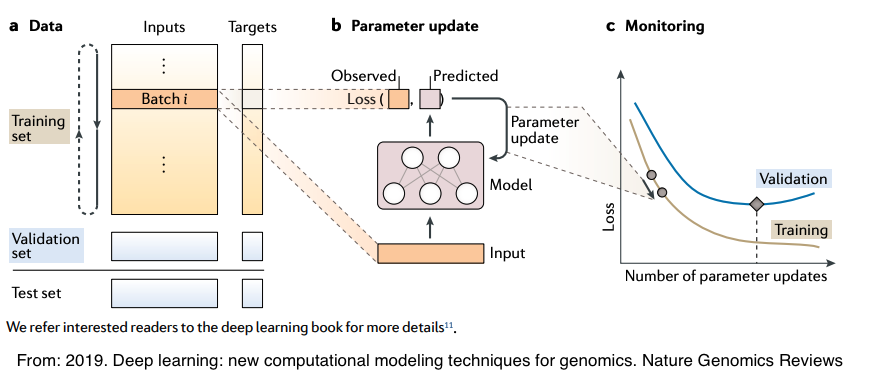
從左邊往右邊來看,左邊是大部分建模前的資料型態,就是一個大表,在監督式學習下,每筆資料都會有個標籤(就是target),通常會把資料切成訓練以及驗證的兩組資料。在以前,建立完建模的資料後,還必須撰寫很複雜的運算,但如今有超多寫得非常簡易的框架,可以直接用,比如下面就是用Python的keras來建立神經網絡的架構:
# this code were from 2019. Deep learning: new computational modelling techniques for genomics. Nature Review Genetics
import keras.layers as kl
from keras.models import Sequential
# Fully connected model architecture
model = Sequential([
k1.Dense(3, activation='relu', input_shape=(2,)),
k1.Dense(2, activation='relu'),
k1.Dense(1, activation='sigmoid')
])
# Specify optimizer, loss and evaluation metric
model.compile(optimizer='adam',
loss='binary_crossentropy',
metrics=['accuracy'])
# Load the dataset
x, y = load_dataset(...)
# Train the model for 10 epochs
model.fit(x,y, epochs=10)
從上面keras的代碼,可以感覺整個撰寫的感受非常好,基本上就像是在寫算式一樣,不太有碼農的味道,反而像是數學式,所以現在這樣的技能快要變成是一種基本配備,這個主題會再往下展開,實在是很有趣,但也很複雜,因為需要理解分子生物學目前已知的現象,然後才有辦法來定義問題,反而問題定義好後,建模的程式代碼已經變得非常親民!
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
目前許多深度學習的相關資源:
閱讀參考:
